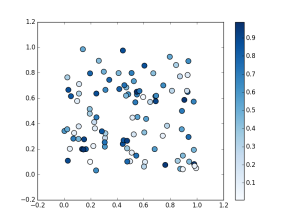
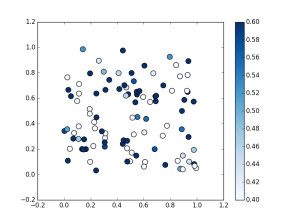
scatter plot by matplotlib
matplotlib.pyplot.scatter の概要
matplotlib には、散布図を描画するメソッドとして、matplotlib.pyplot.scatter が用意されてます。
matplotlib.pyplot.scatter の使い方
matplotlib.pyplot.scatter の主要な引数
| x, y | グラフに出力するデータ |
|---|---|
| s | サイズ (デフォルト値: 20) |
| c | 色、または、連続した色の値 |
| marker | マーカーの形 (デフォルト値: ‘o’= 円) |
| cmap | カラーマップ。c が float 型の場合のみ利用可能です。 |
| norm | c を float 型の配列を指定した場合のみ有効。正規化を行う場合の Normalize インスタンスを指定。 |
| vmin, vmax | 正規化時の最大、最小値。 指定しない場合、データの最大・最小値となります。norm にインスタンスを指定した場合、vmin, vmax の指定は無視されます。 |
| alpha | 透明度。0(透明)~1(不透明)の間の数値を指定。 |
| linewidths | 線の太さ。 |
| edgecolors | 線の色。 |
グラフの出力例
以下例では、100 個 × 2 軸の乱数を2次元座標上にプロットします。
サイズ、色、不透明度、線のサイズ、色を指定
マーカーを指定 (星印)
グラフのタイトル、X 軸、Y 軸の名前 (ラベル)、グリッド線を表示
カラーマップを指定して、値に応じてマーカーを着色
引数 s の値の大小に応じて、色の濃淡やグラデーションで表現することができます。
上記に加えて正規化における最大値 (0.6)、最小値 (0.4) を指定
(右側の凡例の目盛が変わっているのがわかるかと思います)
numpy スライス
pythonのリストやnumpy配列でのスライスは i:j:n の形式で行います。
ここでi は開始インデックス、 j は終了インデックスを表しており、
i以上でjより小さいインデックス(i <= n < j)でスライスされます。
次に n ですが、これはステップ数を表しますが、ステップ数は1の時は省略してi:jのみの記述をすることができます。
ですので質問にある 1:3 は1以上で3より小さいインデックス値 ー (1,2)にスライスされることになります。
また、iやj の値も省略することが可能です。i が省略された場合は i = 0 が設定され、 jが省略された場合はj = (配列のサイズ)が設定されます。
ですので5: インデックス値 5から最後まででスライス:5 最初からインデックス値 4 まででスライス: 最初から最後まで(配列全体)でスライス
ということになります。
最後に、, の説明ですが、numpyでの2次行列の場合 data[行の指定,列の指定]
の記述にて行列から一部をスライスすることができます。
data[:,1:3]は
行は : (行全体)を指定
列は 1:3 インデックス(1,2)を指定
となります。
>>> a = numpy.array([[0,1], [2, 3]])
>>> a
array([[0, 1],
[2, 3]])
>>> a[:,0]
array([0, 2])
>>> [行:列]でスライスでき、省略した場合はすべてを指定したことになるので、[:, 0]は全ての行の0列目を取得することになります。省略せずに書くと2x2の配列の場合ならa[0:2, 0]となります。
scikit-learnで標準化,正規化
標準化の式

正規化の式

scikit-learn で
sklearn の StandardScaler と MinMaxScaler がそれぞれ 標準化 と 正規化 のモジュールです。主に使うメソッドは次の 3 つです。
fit
パラメータ(平均や標準偏差 etc)計算
transform
パラメータをもとにデータ変換
fit_transform
パラメータ計算とデータ変換をまとめて実行
赤:original data
青:標準化データ(standard)
緑:正規化データ(normalized)

code
# coding:utf-8 import matplotlib.pyplot as plt import numpy as np from sklearn.preprocessing import StandardScaler from sklearn.preprocessing import MinMaxScaler # 元データ np.random.seed(seed=1) data = np.random.multivariate_normal( [7, 5], [[5, 0],[0, 2]], 100 ) # 標準化 sc = StandardScaler() data_std = sc.fit_transform(data) # 正規化 ms = MinMaxScaler() data_norm = ms.fit_transform(data) # プロット min_x = min(data[:,0]) max_x = max(data[:,0]) min_y = min(data[:,1]) max_y = max(data[:,1]) plt.figure(figsize=(5, 6)) plt.subplot(2,1,1) plt.title('StandardScaler') plt.xlim([-4, 10]) plt.ylim([-4, 10]) plt.scatter(data[:, 0], data[:, 1], c='red', marker='x', s=30, label='origin') plt.scatter(data_std[:, 0], data_std[:, 1], c='blue', marker='x', s=30, label='standard ') plt.legend(loc='upper left') plt.hlines(0,xmin=-4, xmax=10, colors='#888888', linestyles='dotted') plt.vlines(0,ymin=-4, ymax=10, colors='#888888', linestyles='dotted') plt.subplot(2,1,2) plt.title('MinMaxScaler') plt.xlim([-4, 10]) plt.ylim([-4, 10]) plt.scatter(data[:, 0], data[:, 1], c='red', marker='x', s=30, label='origin') plt.scatter(data_norm[:, 0], data_norm[:, 1], c='green', marker='x', s=30, label='normalize') plt.legend(loc='upper left') plt.hlines(0,xmin=-4, xmax=10, colors='#888888', linestyles='dotted') plt.vlines(0,ymin=-4, ymax=10, colors='#888888', linestyles='dotted') plt.show()
keras モデル構築 評価
#ケース1 score = model.evaluate(x_test, y_test, batch_size=128) #ケース2 scores = model.evaluate(X, Y, verbose=0) print("%s: %.2f%%" % (model.metrics_names[1], scores[1] * 100))
keras モデル構築 学習
#ケース1 data = np.random.random((1000, 100)) labels = np.random.randint(10, size=(1000, 1)) # ラベルデータをカテゴリの1-hotベクトルにエンコードする one_hot_labels = keras.utils.to_categorical(labels, num_classes=10) # 各イテレーションのバッチサイズを32で学習を行なう model.fit(data, one_hot_labels, epochs=10, batch_size=32) #ケース2 # Pima indians datasetをロード dataset = np.loadtxt(os.path.join("..", "data", "pima-indians-diabetes.data"), delimiter=',') # データとラベルを取得 X = dataset[:, 0:8] Y = dataset[:, 8] model.fit(X, Y, nb_epoch=150, batch_size=10) #ケース3 x_train = np.random.random((1000, 20)) y_train = keras.utils.to_categorical(np.random.randint(10, size=(1000, 1)), num_classes=10) model.fit(x_train, y_train, epochs=20, batch_size=128)
keras モデル構築 コンパイル記述
モデルの学習を始める前に,compileメソッドを用いどのような学習処理を行なうかを設定する必要があります.compileメソッドは3つの引数を取ります:
- 最適化アルゴリズム: 引数として,定義されている最適化手法の識別子を文字列として与える(rmspropやadagradなど),もしくは Optimizerクラスのインスタンスを与えることができます. 参考: 最適化
- 損失関数: モデルが最小化しようとする目的関数です.引数として,定義されている損失関数の識別子を文字列として与える(categorical_crossentropyやmseなど),もしくは目的関数を関数として与えることができます.参考:損失関数
- 評価関数のリスト: 分類問題では精度としてmetrics=['accuracy']を指定したくなるでしょう.引数として,定義されている評価関数の識別子を文字列として与える,もしくは自分で定義した関数を関数として与えることができます.
# マルチクラス分類問題の場合 model.compile(optimizer='rmsprop', loss='categorical_crossentropy', metrics=['accuracy']) # 2値分類問題の場合 model.compile(optimizer='rmsprop', loss='binary_crossentropy', metrics=['accuracy']) # 平均二乗誤差を最小化する回帰問題の場合 model.compile(optimizer='rmsprop', loss='mse') # 独自定義の評価関数を定義 import keras.backend as K def mean_pred(y_true, y_pred): return K.mean(y_pred) model.compile(optimizer='rmsprop', loss='binary_crossentropy', metrics=['accuracy', mean_pred])
keras モデル構築 Sequentialモデルでの構築
Sequentialモデルでの構築
#.addで組む際はこのやり方 model = Sequential() model.add(Dense(12, input_dim=8, init='uniform', activation='relu')) model.add(Dense(8, init='uniform', activation='relu')) model.add(Dense(1, init='uniform', activation='sigmoid')) #input_dim それぞれの入力blobのdim、各blobはnum, channels, height, widthの4つの次元を持つ。
- initで層の重みの初期化方法を指定できる
- uniformだと0~0.05の一様乱数。normalだと正規乱数。Deep Learning Tutorialの初期値重みで使われていたglorot_uniformもある層の活性化関数は、独立した層ではなくDenseのactivation引数でも指定できる
- 隠れ層の活性化関数にはrelu、出力層の活性化関数にはsigmoidを指定
- 出力層にsigmoidを使うと0.0から1.0の値が出力されるため入力データのクラスが1である確率とみなせる。0.5を閾値として0.5未満ならクラス0、0.5以上ならクラス1として分類する.
他に例えば
入力層784次元、中間層1024次元、出力層10次元のニューラルネットワークの構築は
model = Sequntial() model.add(Dense(1024, input_dim=784)) model.add(Activation('sigmoid')) model.add(Dense(10)) model.add(Activation('softmax'))
モデル:例2
model = Sequential( model.add(Dense(512, input_shape=(784,))) model.add(Activation('relu')) model.add(Dropout(0.2)) model.add(Dense(512)) model.add(Activation('relu')) model.add(Dropout(0.2)) model.add(Dense(10)) model.add(Activation('softmax')) return model

KerasのConvolution2Dを使う時にborder_modeという引数があり、'valid'と'same'が選択できるのですが、これが何なのかを調べるとStackExchangeに書いてありました(convnet - border_mode for convolutional layers in keras - Data Science Stack Exchange)。
‘valid’
出力画像は入力画像よりもサイズが小さくなる。

‘same’
ゼロパディングすることで、出力画像は入力画像と同じサイズになる。

ゼロパディングすることのメリットについてはここ(定番のConvolutional Neural Networkをゼロから理解する - DeepAge)に書いてあります。
端のデータに対する畳み込み回数が増えるので端の特徴も考慮されるようになる
畳み込み演算の回数が増えるのでパラメーターの更新が多く実行される
カーネルのサイズや、層の数を調整できる